青竹调,萨萨拉比姆,电脑维修
chapter2 which dna patterns play the role of molecular clocks
寻找模序
一、
转录因子会结合基因上游的特定序列,调控基因的转录表达,但是在不同个体中,这个序列会有一些差别。本章讲述用贪婪、随机算法来寻找这个序列:寻找模序。

二、一些概念:
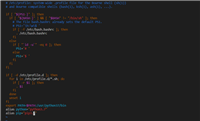
1. score、profile 的含义如图
根据profile matrix 可以计算出某个kmer在某一profile下的概率

三、
提出问题:motif finding problem:
given a collection of strings, find a set of k-mers, one from each string, that minimizes the score of the resulting motif.
input: a collection of strings dna and an integer k.
output: a collection motifs of k-mers, one from each string in dna, minimizing score(motifs) among all possible choices of k-mers.
一组序列中,寻找一组k-mer,它们的score是最低的(或者与consensus sequence的海明距离之和最小)
1 遍历
medianstring(dna, k)
distance ← ∞
for each k-mer pattern from aa…aa to tt…tt
if distance > d(pattern, dna)
distance ← d(pattern, dna)
median ← pattern
return median
2 贪婪法 greedymotifsearch
greedymotifsearch(dna, k, t)
bestmotifs ← motif matrix formed by first k-mers in each string
from dna
for each k-mer motif in the first string from dna
motif1 ← motif
for i = 2 to t
form profile from motifs motif1, …, motifi - 1
motifi ← profile-most probable k-mer in the i-th string
in dna
motifs ← (motif1, …, motift)
if score(motifs) < score(bestmotifs)
bestmotifs ← motifs
output bestmotifs
详解 http://www.mrgraeme.co.uk/greedy-motif-search/
*贪婪法 greedymotifsearch with pseudocounts
pseudocounts:在形成profile matrix时,给0项设为一个较小的值
greedymotifsearch(dna, k, t)
form a set of k-mers bestmotifs by selecting 1st k-mers in each string from dna
for each k-mer motif in the first string from dna
motif1 ← motif
for i = 2 to t
apply laplace's rule of succession to form profile from motifs motif1, …, motifi-1
motifi ← profile-most probable k-mer in the i-th string in dna
motifs ← (motif1, …, motift)
if score(motifs) < score(bestmotifs)
bestmotifs ← motifs
output bestmotifs
3. 随机法randomized motif search
randomizedmotifsearch(dna, k, t)
#随机从每个dna取k-mer,生成一组motifs randomly select k-mers motifs = (motif1, …, motift) in each string from dna bestmotifs ← motifs while forever profile ← profile(motifs)#根据motifs形成profile矩阵 motifs ← motifs(profile, dna) #根据profile矩阵从一组dna生成一组几率最大的motifs if score(motifs) < score(bestmotifs) bestmotifs ← motifs else return bestmotifs
随机算法起到作用的原因是,随机选取的一组motifs,有可能选到潜在正确的一个k-mer,那么就在这中形成倾斜,直至寻找到较优解
改进: 上一个算法,每次迭代都重新随机生成一组新的motifs,这可能把潜在的正确模序抛弃了,改进的方法是每次随机只更改一行k-mer

gibbssampler(dna, k, t, n)
randomly select k-mers motifs = (motif1, …, motift) in each string from dna
bestmotifs ← motifs
for j ← 1 to n
i ← random(t)
profile ← profile matrix constructed from all strings in motifs except for motif[i]
motif[i] ← profile-randomly generated k-mer in the i-th sequence
if score(motifs) < score(bestmotifs)
bestmotifs ← motifs
return bestmotifs
如对本文有疑问,请在下面进行留言讨论,广大热心网友会与你互动!! 点击进行留言回复

Python 实现将numpy中的nan和inf,nan替换成对应的均值
python爬虫把url链接编码成gbk2312格式过程解析

网友评论